-
@@ -870,7 +852,7 @@
+
-
-
-
- 本篇
-
+ 上一篇tr -d '\r' ## 删除 \r
sed
sed 's/^M$//g' filename > tmp_filename使用sed 工具:需要注意打^M时应输入Ctrl+v+m,长按Ctrl依次点击v和m键,而不是数字6上的^符号。
reference
使用sed 工具:需要注意打^M时应输入Ctrl+v+m,长按Ctrl依次点击v和m键 (或者输入Ctrl+V,然后输入Ctrl+M),而不是数字6上的^符号。
reference
dos2unix (if installed)
 +
+  - wetlab-spCas9KO
+ wetlab-CoIPw
- wetlab-spCas9KO
+ wetlab-CoIPw
 +
+ Bioinfo.multi-sequence-align
+
+
+ Bioinfo.multi-sequence-align
+  +
+ wetlab-spCas9KO
+
+
+ wetlab-spCas9KO
+ 在研究一个基因时,常常会需要了解这个基因的(核苷酸或氨基酸)序列在物种间的保守性。下面提供一种查询基因同源情况、序列比对及可视化的方法。以基因 SF3B1 为例:
+如果是well-known的基因,保守性大概了解,可以自行判断是使用核苷酸序列(非常保守时使用)还是氨基酸序列(不怎么保守时使用)。
+如果未知,可以通过以下途径知晓:
+如果你想显示有哪些物种,可以使用TimeTree绘制指定物种间的进化树。
+确定要比较的物种,获取这些物种SF3B1的序列,用 snapGene 进行多序列比对。
多比对的结果 export为fasta格式。
!!!如果比对结果中,个别地方有碱基的错位,可手动编辑fasta文件,进行修改。
+下面提供一个在线的简单可视化工具:碱基顺序比对工具
+将比对结果fasta文件输入后,submit,出现的结果,可点击鼠标右键,直接打印输出为PDF文件。或者复制存储为其他类型文件。
+待更新…
+ + + +
+ Bioinfo.multi-sequence-align
+
+
+ Bioinfo.multi-sequence-align
+  +
+ wetlab-CoIPw
+
+
+ wetlab-CoIPw
+  +
+ PaperRead-Day14
+
+
+ PaperRead-Day14
+  -
- 文献阅读 Day6
-
-
- 文献阅读 Day6
-  -
- 文献阅读 Day5
-
-
- 文献阅读 Day5
-  +
+ 文献阅读 Day6
+
+
+ 文献阅读 Day6
+  +
+ 文献阅读 Day5
+
+
+ 文献阅读 Day5
+  -
- R数据科学,第一部分:探索
-
-
- R数据科学,第一部分:探索
-  -
- RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
-
-
- RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
-  +
+ R数据科学,第一部分:探索
+
+
+ R数据科学,第一部分:探索
+  +
+ RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
+
+
+ RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
+  +
+ Bioinfo.multi-sequence-align
+
+
+ Bioinfo.multi-sequence-align
+  +
+ wetlab-CoIPw
+
+
+ wetlab-CoIPw
+  -
- Docker-入门
-
-
- Docker-入门
-  -
- R 脚本编写指南
-
-
- R 脚本编写指南
-  +
+ Docker-入门
+
+
+ Docker-入门
+  +
+ R 脚本编写指南
+
+
+ R 脚本编写指南
+  -
- Bioinfo-RoutineToolsCollections
-
-
- Bioinfo-RoutineToolsCollections
-  -
- SNV 数据搜集与关联分析
-
-
- SNV 数据搜集与关联分析
-  +
+ Bioinfo-RoutineToolsCollections
+
+
+ Bioinfo-RoutineToolsCollections
+  +
+ SNV 数据搜集与关联分析
+
+
+ SNV 数据搜集与关联分析
+ 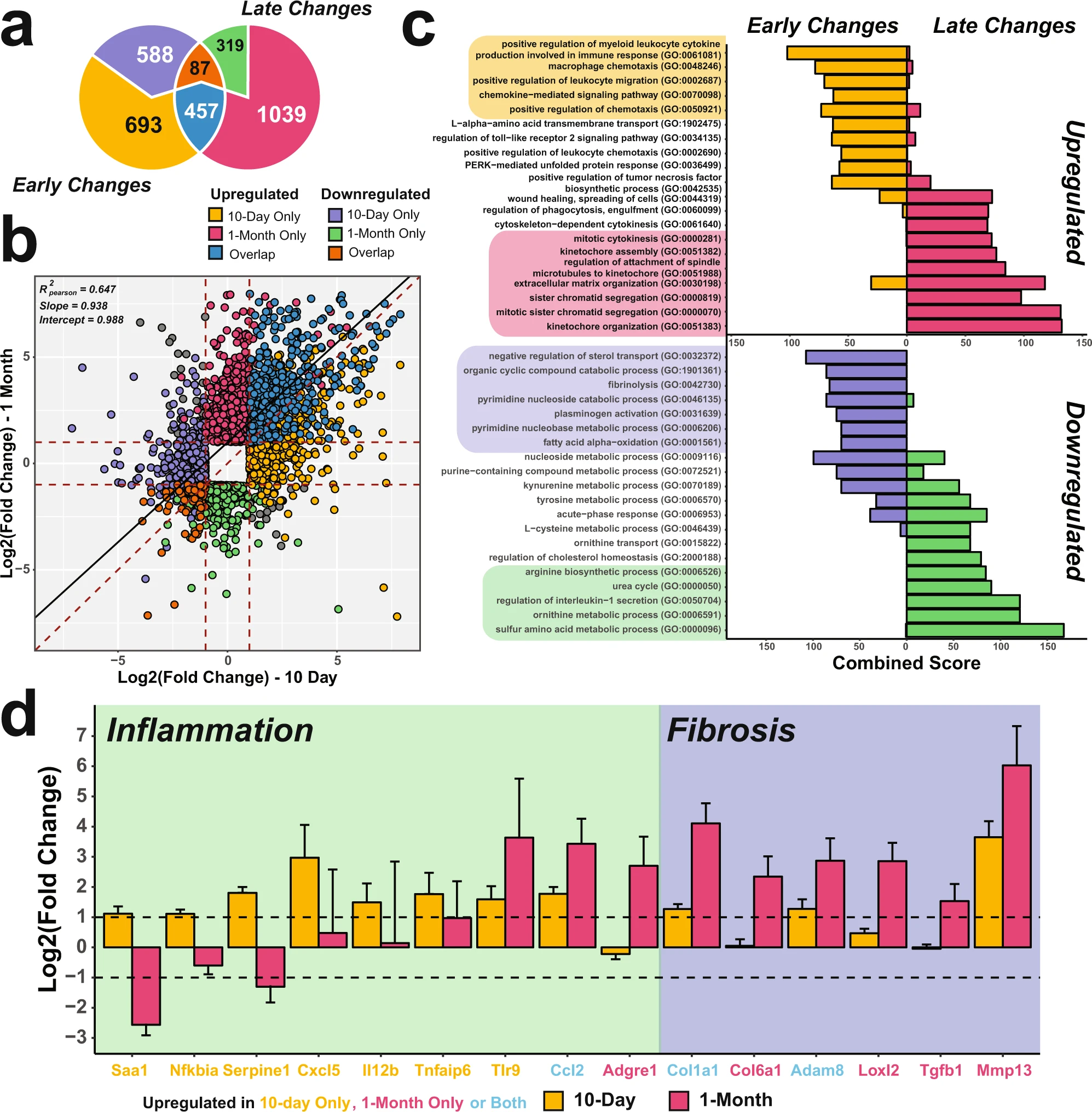 -
- 基因富集分析
-
-
- 基因富集分析
-  -
- R 数据科学:数据加工与重塑
-
-
- R 数据科学:数据加工与重塑
-  +
+ 基因富集分析
+
+
+ 基因富集分析
+  +
+ R 数据科学:数据加工与重塑
+
+
+ R 数据科学:数据加工与重塑
+  -
- linux命令——expr
-
-
- linux命令——expr
-  -
- linux命令——grep
-
-
- linux命令——grep
-  +
+ linux命令——expr
+
+
+ linux命令——expr
+  +
+ linux命令——grep
+
+
+ linux命令——grep
+  -
- 文献阅读 Day15
-
-
- 文献阅读 Day15
-  -
- PaperRead-Day14
-
-
- PaperRead-Day14
-  +
+ 文献阅读 Day15
+
+
+ 文献阅读 Day15
+  +
+ PaperRead-Day14
+
+
+ PaperRead-Day14
+  -
- 文献阅读 Day6
-
-
- 文献阅读 Day6
-  -
- 文献阅读 Day5
-
-
- 文献阅读 Day5
-  +
+ 文献阅读 Day6
+
+
+ 文献阅读 Day6
+  +
+ 文献阅读 Day5
+
+
+ 文献阅读 Day5
+  -
- R数据科学,第一部分:探索
-
-
- R数据科学,第一部分:探索
-  -
- RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
-
-
- RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
-  +
+ R数据科学,第一部分:探索
+
+
+ R数据科学,第一部分:探索
+  +
+ RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
+
+
+ RNA 剪接因子 SRRM3 和 SRRM4 能够作为去势抵抗性的神经内分泌性前列腺癌的分子表征
+